Link Communities
| Kevin Schwarz | Lennard Heller | Xuan Thanh Nguyen | Minh Vu Ngo |
| RWTH Aachen | RWTH Aachen | RWTH Aachen | RWTH Aachen |
| kevin.schwarz@rwth-aachen.de | lennard.heller@rwth-aachen.de | xuan.thanh.nguyen@rwth-aachen.de | vu.ngo@rwth-aachen.de |
Um Systeme von interagierenden Objekten wie zum Beispiel den biologischen Organismus, menschliches Sozialleben oder auch das soziale Netzwerk von Handybenutzern besser verstehen zu können, ist das Bilden von Netzwerken einer der wichtigsten Ansätze geworden. Dabei wird versucht Communities in einem Netzwerk zu identifizieren, welche nicht direkt voneinander abgegrenzt sein müssen. Ein Knoten eines Netzwerkes kann sich gleichzeitig in verschiedenen Untergruppen befinden. Des Weiteren sind Communities oftmals hierarchisch aufgebaut, wodurch ihre Struktur ebenso hierarchisch aufgebaut wird. Die Verbindung der Untergruppen und der hierarchischen Struktur lässt uns die Bildung von übergreifenden Gruppen nur sehr schwer oder meist auch gar nicht ermöglichen. Daher werden in diesem Ansatz die Verbindungen der Communities und nicht die Knoten an sich betrachtet. Es wird gezeigt, dass die entgegengesetzten Strukturen der Hierarchie und überschneidenden Communities vereint werden können. Dazu werden Link Communities anhand von einigen Beispielen betrachtet und Algorithmen vorgestellt, die die Qualität der zugehörigen Cluster bewerten. Es wird gezeigt, dass überschneidende und hierarchische Organisation in Netzwerken zwei Aspekte desselben Phänomens sind.
1. Einleitung
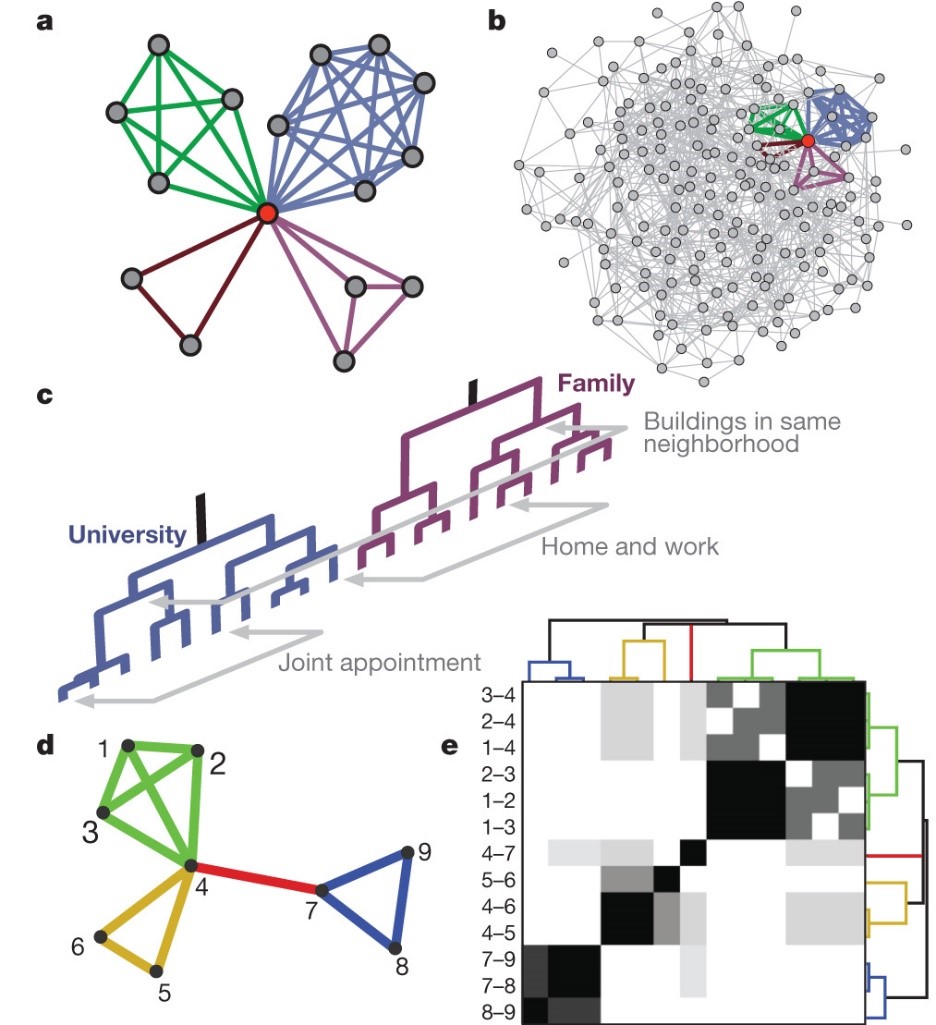
Obwohl es keine allgemein anerkannte Definition gibt, ist es weitgehend Konsens, dass eine Community mehr interne als externe Verbindungen haben sollte.(Radicchi et al., 2004) (Newman, Mark E. J. & Girvan, 2004) (Rosvall & Bergstrom, 2008) (Reichardt & Bornholdt, 2004) (Li, 2008) (Lancichinetti & Fortunato, 2009) Im Gegensatz zur eigenen Intuition jedoch können sich überschneidende Communities deutlich mehr externe als interne Verbindungen haben (Abb. 1a, b). Da sich durchgängig überschneidende Communities sogar in diesen grundlegenden Annahmen widersprechen, ist eine neue Herangehensweise notwendig. Die Entdeckung der Hierarchie und der Organisation von Communities wurde schon immer als Problem des Festlegens der richtigen Zugehörigkeit jedes Knotens betrachtet. Anzumerken ist, dass während Knoten zu mehreren Gruppen gehören (Einzelpersonen haben Familien, Mitarbeiter und Freunde, Abb. 1c), existieren Links aus einem dominanten Grund (zwei Personen sind in der gleichen Familie, arbeiten zusammen oder haben gemeinsame Interessen). Anstatt anzunehmen, dass eine Community eine Menge an Knoten mit vielen Links (bzw. Verbindungen) zwischen ihnen ist, betrachten wir eine Community als eine Menge von eng zusammenhängenden Links. Jeden Link in einem individuellen Kontext zu betrachten, erlaubt es uns hierarchische und sich überschneidende Beziehungen gleichzeitig aufzuzeigen.
Wir benutzen hierarchische Clusterbildung mit Ähnlichkeit zu Links, um ein Dendrogramm zu erstellen, bei dem jedes Blatt ein Link des originalen Netzwerkes und Äste die Link Communities darstellen (Abb. 1d, e und Methoden). In diesem Dendrogramm besetzen Links einzigartige Positionen, wohingegen Knoten naturgemäß mehrere Positionen besetzen, die sie ihren Links zu verdanken haben. Wir extrahieren Link Communities auf vielen Ebenen indem wir dieses Dendrogramm an verschiedenen Grenzbereichen abschneiden. Jeder Knoten trägt alle Zugehörigkeiten seines Links in sich und kann daher zu verschiedenen, sich überschneidenden Communities gehören. Obwohl wir nur eine einzige Zugehörigkeit je Link zuteilen, können Link Communities auch viele Beziehungen zwischen Knoten erfassen, weil verschiedene Knoten gleichzeitig zu mehreren Communities gehören können. Das Link Dendrogramm bietet eine vielfältige Strukturhierarchie, aber um die relevantesten Communities zu erhalten, ist es notwendig die beste Stelle, an der man den Baum abschneidet, zu bestimmen. Aus diesem Grund führen wir eine natürliche Zielfunktion ein, die Abtrennungsdichte D, basierend auf der Linkdichte innerhalb der Communities. Im Gegensatz zum Bausteinprinzip (Newman, Mark E. J. & Girvan, 2004) leidet D nicht unter einer Auflösungsgrenze (Fortunato & Barthélemy, 2007) (Methoden). Die Berechnung von D auf jeder Ebene des Link Dendrogramms erlaubt es uns, die beste Ebene zum Abschneiden herauszusuchen (obwohl auch eine bedeutende Struktur über und unter dieser Schwelle existiert). Es ist auch möglich D direkt zu optimieren. Wir können nun die Entdeckung sich überschneidender Communities, sowie gut gestellte Optimierungsprobleme ausformulieren, um jede Überschneidung erklären zu können ohne beachten zu müssen, dass Knoten Teil vieler Communities sind.
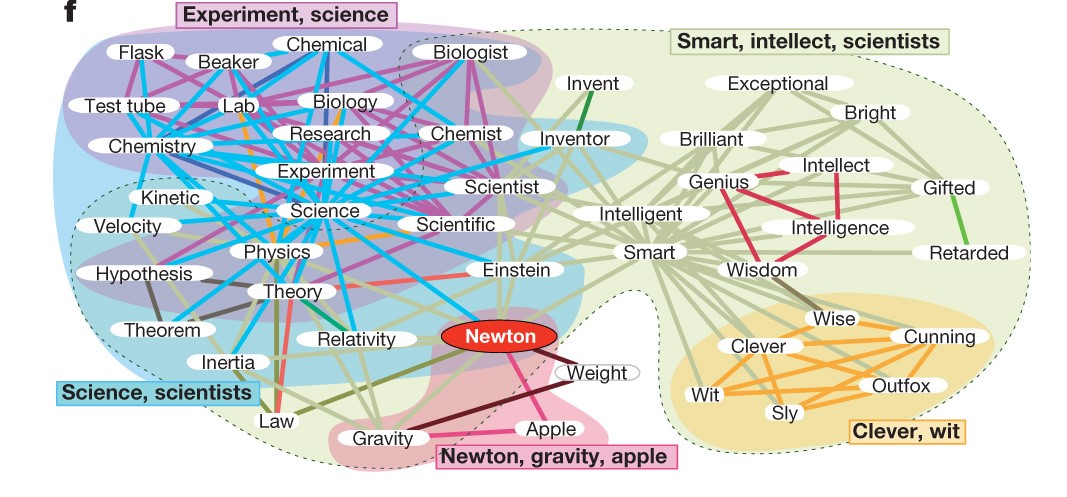
Als erklärendes Beispiel zeigt Abb. 1f Link Communities um das Wort „Newton“ in einem Netzwerk von üblicherweise assoziierten englischen Wörtern. (siehe zusätzliche Informationen, Teil 6, für Einzelheiten über Netzwerke, die im Verlauf des Textes benutzt werden.) Die „clever, Verstand“ Community wird korrekterweise innerhalb der „schlau/Intellekt“ Community ausfindig gemacht. Die Worte „Newton“ und „Schwerkraft“ gehören beide in die „schlau/Intellekt“, „Gewicht“ und „Apfel“ Community, um zu illustrieren, dass Link Communities viele Beziehungen zwischen Knoten erfassen.
|
|
2. Verwandte Arbeiten
Diese Arbeit baut größtenteils auf dem Paper „Link communities reveal multiscale complexity in networks“ von Y. Ahn, J. Bagrow und S. Lehmann auf. (Yong-Yeul et al., 2010) Mit einer auf Java basierten Peer-to-Peer Infrastuktur werden
5 weiteren OCD-Algorithmen(Overlapping Community Detection) verglichen. Die Ergebnisse werden in einem späteren Teil dieser Arbeit dargestellt.
Eine vergleichbare Herangehensweise wurde von T. S. Evans und R. Lambiotte entwickelt. (Evans & Lambiotte, 2009) (Evans T.S. & Lambiotte R., 2009)
3. Methodik
Link Communities
Für ein ungerichtetes, ungewichtetes Netzwerk bezeichnen wir die Menge der Knoten \(i\) und deren Nachbarn als \(n_+ (i)\).
Wir betrachten nur die Verbindungspaare, die sich einen Knoten teilen, da wir uns von diesen erhoffen, dass sie mehr
Ähnlichkeiten aufweisen als Verbindungspaare, deren Knoten voneinander getrennt sind. Wir definieren die Ähnlichkeit
\(S\) zwischen den Verbindungen \(e_{ik}\) und \(e_{jk}\) wie folgt:
Der geteilte Knoten \(k\) erscheint nicht mehr in S, da dieser keine weiteren Informationen enthält. Aus der Gleichung \((1)\) bildet nun ein Dendrogramm. Wenn man dieses Dendrogramm an bestimmen Stellen,wie zum Beispiel an der größten Partitionsdichte, teilen, erhalten wir Link Communities.
Partitionsdichte
Für ein Netzwerk mit \(M\) Verbindungen und \(N\) Knoten, sei \(P={P_1,…,P_C }\) eine Partition aus Verbindungen in \(C\) Teilmengen.
Die Anzahl der Verbindungen in der Teilmenge \(P_c\) beträgt \(m_c=|P_c |\). Die Anzahl der induzierten Knoten, alle Knoten, die
diese Verbindung berühren, beträgt \(n_c=|\bigcup\limits_{e_{ij} \in P_{i}} {i,j}|\) Beachte, dass \(\sum_\limits{c}m_{c}=M\) und \(\sum_\limits{c}n_{c} \ge N\) (vorausgesetzt
sind keine isolierten Knoten). Die Verbindungsdichte \(D_{c}\) einer Community \(c\) ist definiert durch:
Dies ist die Anzahl an Verbindungen in \(P_{c}\) normalisiert nach der minimalen und maximalen Anzahl von möglichen Verbindungen zwischen diesen Knoten, vorausgesetzt sie sind verbunden (Wir setzten voraus, dass \(D_{c}=0\), falls \(n_{c}=2\)). Die Partitionsdichte \(D\) ist der Durchschnitt der \(D_{c}\), gewichtet mit folgenden Verbindungen:
\[D = \frac{2}{M} \sum\limits_{c} m_{c}\frac{m_{c}-(n_c-1)}{(n_c-2)(n_c-1)}\qquad \qquad \qquad(2)\]Gleichung \((2)\) besitzt keine Auflösungsgrenze (Fortunato & Barthélemy, 2007) , da jeder Term lokal in \(c\) ist.
Community Validierung
Vorausgesetzt ist eine nicht-triviale Community mit mind. 3 Knoten. Wir benutzen die Metadaten der „Anreicherung“, um die Qualität
der Communities, bezüglich der Ähnlichkeit der Knoten zu nicht-trivialen Communities in Relation zu allen Knoten zu beurteilen
(engl.: global baseline). „Overlap quality“ ist die gegenseitige Information über die Anzahl der nicht-trivialen Mitgliedschaften und der
übergreifenden Metadaten. „Community Abdeckung“ (engl.: Community coverage) ist die Einteilung von Knoten, die zu mehr als einer nicht-trivialen Community gehört.
Weil Methoden mit gleicher Community Abdeckung eine andere Anzahl von Überdeckungen ergeben können, ist die „Überschneidungs Abdeckung“ (engl.: Overlap coverage) die
durchschnittliche Anzahl von nicht-trivialen Mitgliedschaften pro Knoten (äquivalent zu Community Abdeckung für nicht überlappende Methoden).
Dendrogramm
Um die Bildung von Clustern gut darstellen zu können, werden oft Dendrogramme verwendet (bspw. in Abbildung 1c zu sehen). Dendrogramme sind Baumdiagramme,
die schrittweise einzelne Elemente (in unserem Fall Paare von Kanten) oder auch Cluster vereinen. Dabei ist die Wurzel des Baumes ein einziges Cluster und
die Blätter sind einzelne Elemente. Des Weiteren besitzt jede Kante noch ein Attribut, welches mit Abstand bezeichnet wird.
Der Vorteil dieser Darstellung liegt darin, dass man die Abstände der Vereinigung der Elemente/Cluster mithilfe von Vertikalen oder auch
Horizontalen gut darstellen kann. Als Abstände werden in unseren Dendrogrammen die unterschiedlichen Ähnlichkeiten \(S(e_{ij},e_{jk})\) verwendet.
Kontroll Dendrogramm
Um zu testen, ob eine hierarchische Struktur bei einer bestimmten Schwelle, \(t_{*}\), korrekt ist, führen wir folgende Kontrolle ein:
Als erstes berechnen wir die Ähnlichkeiten \(S(e_{ij},e_{jk})\) für alle verbundenen Kantenpaare \((e_{ik},e_{jk})\) wie gewöhnlich. Dann führen
wir unser hierarchisches single-linkage Clustering durch, und vereinen alle Kantenpaare absteigend in \(S\) für \(S \ge t_{*}\) und bauen
die Community Struktur bis \(t=t_{*}\) auf. Unter \(t_{*}\) mischen wir zufällige Ähnlichkeiten der übrigen Kantenpaare mit \(S<t_{*}\) und führen dann
das Vereinen wie zuvor aus. Das zufällige Auswählen ändert nur die Reihenfolge der Vereinigungen und stellt sicher, dass die Kantenpaare,
die vereint werden, gleichbleiben, da dieselben Ähnlichkeiten gruppiert werden. Dies bestimmt nicht nur die Geschwindigkeit der
Vereinigungen, sondern auch die Verteilung der Ähnlichkeiten und die stark ausgeprägte Community Struktur, die in \(t_{*}\) gefunden wurde. Dieses
Verfahren stellt sicher, dass das Dendrogramm zufällig erstellt wird, wobei hervorstechende Eigenschaften erhalten bleiben.
4. Anwendung
Die Autoren Y. Ahm, J. Bagrow und S. Lehmann wollen zeigen, dass eine link-basierte Herangehensweise der bereits existierenden knoten-basierten Herangehensweise überlegen ist. Dazu vergleichen sie LC mit 3 Methoden, die konzipiert wurden, um knoten-basierte Communities zu finden. Diese Methoden sind Gruppen Filtration (Palla et al., 2007) (engl.: clique percolation), Greedy Modularitätsoptimierung (Clauset et al., 2004) (engl.: greedy modularity optimization) und Infomap (Rosvall & Bergstrom, 2008).
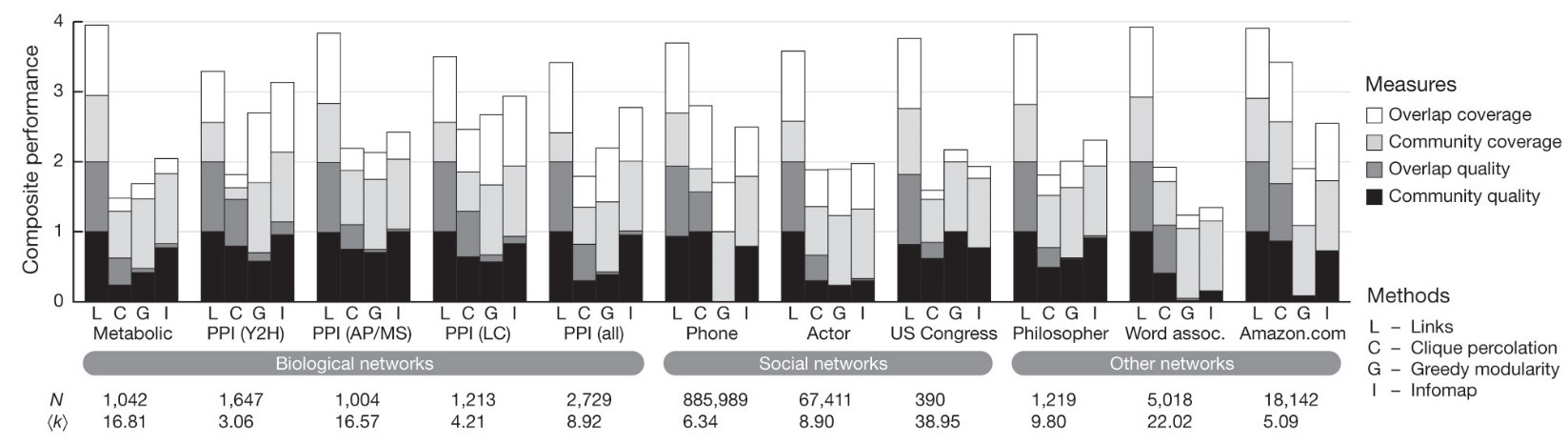
Die 4 Algorithmen werden auf eine Testgruppe von 11 Netzwerken, unter anderem das Mobilfunknetz, das metabolische Netzwerk iAF1260 vom Escherichia colli K-12 MG1655 Erregerstamm und Netzwerke der Protein-Protein Interaktion, angewendet und die Ergebnisse miteinander verglichen. Der Vergleich betrachtet die 4 Kriterien: Übereinstimmung der Communities, Übereinstimmung der Überschneidungen, Qualität der Communities und Qualität der Überschneidungen.
Wie in Abbildung 2 zu sehen, zeigt der Vergleich, dass LC in allen Aspekten besser abschneidet als seine Kontrahenten. Daraus resultierend kommen Y. Ahm, J. Bagrow und S. Lehmann zu dem Ergebnis, dass LC bzw. die link-basierte Herangehensweise der knoten-basierten Herangehensweise überlegen ist.
Im Folgenden vergleichen wir Link Communities mit 5 anderen OCD-Algorithmen: CLIZZ, DMID, MONC, SLPA und SSK. Die Algorithmen werden auf verschiedene Graphen, die mit dem Newman- oder LFR-Verfahren erzeugt wurden, getestet. Der Vergleich erfolgt anhand folgender Kriterien:
- Laufzeit
- Omega Index: Kriterium für die Übereinstimmung der gefundenen Communities mit den tatsächlichen Communities
- Extended Normalized Mutual Information (ENMI): zusätzliches Kriterium für die Übereinstimmung der gefundenen Communities mit den tatsächlichen Communities
- Modularität
Graph mit 128 Knoten, 2048 Kanten und 4 Communities (Newman):
Ursprünglich sollten die Algorithmen auf 4 Graphen getestet werden. Da die Laufzeit von Link Communities jedoch sehr hoch ist, konnten die Tests für Graphen mit mehr als 500 Knoten nicht abgeschlossen werden.
Das Netzdiagramm zeigt, dass der Algorithmus zur Berechnung der Link Communities die mit Abstand schlechteste Laufzeit von allen betrachteten OCD-Algorithmen besitzt. In Hinsicht auf den Omega-Index schneidet Link Communities durchschnittlich ab und bei den Kategorien „Modularität“ und „ENMI“ konnte jeweils der zweite Platz erreicht werden.
Aus diesem Vergleich lässt sich kein Fazit über die Qualität von Link Communities ziehen, da ein Graph nicht repräsentativ für die Allgemeinheit ist. Es lässt sich jedoch sagen, dass die Vergleiche von den Entwicklern von Link Communities und die daraus resultierenden Ergebnisse wichtige Kriterien, wie die Laufzeit, für einen guten Algorithmus nicht berücksichtigen.
5. Zusammenfassung
Viele moderne Netzwerke sind weit davon entfernt, komplett zu sein. Dem ambitionierten Projekt zur Einzeichnung aller Protein-Protein Interreaktionen in Hefe etwa, wird momentan die Entdeckung von ungefähr 20% aller Verbindungen (Yu, 2008) zugetraut. Durch die Erhöhung der Datensammelrate werden Netzwerke immer dichter und dichter, Überschneidungen werden immer verbreiteter und Herangehensweisen, welche speziell zum Entwirren komplexer und sich stark überschneidender Strukturen entwickelt werden, werden immer essenzieller. Allgemein gesagt steht der Perspektivwechsel von Knoten zu Links stellvertretend für einen fundamental neuen Weg, komplexe Systeme zu untersuchen. An dieser Stelle haben wir einen Schritt vorwärts gemacht, um die Konsequenzen einer link-basierten Herangehensweise zu verstehen. Jedoch haben wir in unseren Testgraphen auch gesehen, dass der vorgestellte Algorithmus zur Erkennung von Link Communities in der Praxis nicht anwendbar ist, da die Laufzeit viel zu hoch ist. Hier gibt es noch großes Potential zur Optimierung des Alogrithmus.
6. Referenzen
- Radicchi, F., Castellano, C., Cecconi, F., Loreto, V., & Parisi, D. (2004). Defining and identifying communities in networks. PROCEEDINGS OF THE NATIONAL ACADEMY OF SCIENCES OF THE UNITED STATES OF AMERICA, 101(9), 2658–2663. https://doi.org/10.1073/pnas.0400054101
- Newman, Mark E. J., & Girvan, M. (2004). Finding and Evaluating Community Structure in Networks. Physical Review, 69(026113).
- Rosvall, M., & Bergstrom, C. T. (2008). Maps of random walks on complex networks reveal community structure. Proceedings of the National Academy of Sciences, 105(4), 1118–1123. https://doi.org/10.1073/pnas.0706851105
- Reichardt, J., & Bornholdt, S. (2004). Detecting fuzzy community structures in complex networks with a Potts model. Physical Review Letters, 93(21). https://doi.org/10.1103/PhysRevLett.93.218701
- Li, D. (2008). Synchronization interfaces and overlapping communities in complex networks. Phys Rev Lett, 101(168701).
- Lancichinetti, A., & Fortunato, S. (2009). Community detection algorithms: A comparative analysis. PHYSICAL REVIEW E, 80(5). https://doi.org/10.1103/PhysRevE.80.056117
- Fortunato, S., & Barthélemy, M. (2007). Resolution limit in community detection. PROCEEDINGS OF THE NATIONAL ACADEMY OF SCIENCES OF THE UNITED STATES OF AMERICA, 104(1), 36–41. https://doi.org/10.1073/pnas.0605965104
- Yong-Yeul, A., Bagrow, J. P., & Lehmann, S. (2010). Link communities reveal multiscale complexity in networks. Nature, 09182.
- Evans, T. S., & Lambiotte, R. (2009). Line graphs, link partitioms and overlapping communities. Phys. Rev. E, 80(016105).
- Evans T.S., & Lambiotte R. (2009). Edge partitions and overlapping communities in complex networks. http://arxiv.org/abs/0912.4389
- Palla, G., Barabási, A.-L., & Vicsek, T. (2007). Quantifying social group evolution. Nature, 446(7136), 664–667. https://doi.org/10.1038/nature05670
- Clauset, A., Newman, Mark E. J., & Moore, C. (2004). Finding community structure in very large networks. Phys. Rev., E 70(6).
- Yu, H. (2008). High-quality binary protein interaction map of the yeast interactome network. Science, 322, 104–110.